Haldja Viinalass, Sirje Värv
Eesti Maaülikooli veterinaarmeditsiini ja loomakasvatuse instituut
Ilmus ajakirjas „Tõuloomakasvatus“ 3/2021
Tori hobusetõug on välja kujunenud keeruka uudikristamise teel – kohaliku Eesti hobuse parandamine viis uue hobusetõu tunnustamiseni. Erinevalt näiteks eesti hobuse aretusest, kus tõu arengu erinevatel etappidel on sisestavaks ristamiseks kasutatud paari importtõugu (soome hobune, araabia täisvereline), on tori tõu aretuses kasutatud arvukalt importtõuge.
Tori hobuse tõuraamat on jagatud osadeks hobuste põlvnemisandmete alusel. Viimasel kümnendil on seoses tori tõuga mitmeid kordi tõstatunud küsimused: kas tõus on eristuvaid alampopulatsioone, juhul kui alampopulatsioonid on olemas, siis kui kaugel üksteisest on alampopulatsioonid geneetiliselt, kas tori tõu alampopulatsioonid on omavahel geneetiliselt sarnased või on mõni alampopulatsioon kaugenenud ja sarnaneb enam mõne teise Eestis aretatava kohaliku hobuse tõuga või Eestis kasvatatava rahvusvahelise tõu populatsiooniga või on see üldse teistest alampopulatsioonidest ning eelnimetatud tõugudest eristunud.
Käesoleva artikli eesmärgiks on tutvustada peamisi tulemusi sel aastal valminud tori hobusetõu uuringust koos viidetega varasematele uuringutele, mis võiks tori tõu kujunemist selgitada ja olla abiks tõu säilitamise korraldamisel.
Tori hobuse tõug on geneetliselt mitmekesine
Varasematest geneetiliste markerite alusel läbi viidud uuringutest (DNA mikrosatelliidid ja mitokondri DNA; Sild et al., 2019a, 2019b) on teada, et tori tõug on geneetiliselt mitmekesine. Ka põlvnemisandmetest lähtub, et tõu genofondi on valiku kõrval mõjutanud sissetoodud geneetiline materjal – eelkõige imporditud täkud peamiselt soojaverelistest tõugudest. Kokku kajastus sugupuudes erinevaid tõuge > 30 (2013. a tori uuringu lõpparuanne). Sagedamini oli aastail 1870-2012 sündinud hobuste põlvnemistabelites leida postjee-bretooni, hannoveri, ida-friisi, trakeeni, holsteini ja inglise täisverelisest hobusetõust pärit eellasi ning tori tõu algkomponente – eesti ja norfolki-roadsteri hobust. Praegu kehtiva aretusprogrammi järgi on neist tori tõugu kujundanud tõugudest lubatud vanempaare kombineerida tori ja hannoveri, trakeeni, holsteini ning inglise täisverelise hobusetõuga, st ristamisi nendest komponenttõugudest täkkudega, kuid see puudutab vaid aretussuuna hobuseid (tõuraamatu TB-osa). 2013. a läbiviidud tori tõu uuringu materjalidest on näha, et märadest on läbi kogu tõu ajaloo muu tõukuuluvusega olnud väga väike osa, kuid üsna palju on mõnel etapil (nt tõuaretuse algusaastail ja 1940ndail) suguhobuste emadest märgitud ristandite ja päritolult mitte teada olevaina.
Emapoolse geneetilise päritolu kohta on võimalik täpsemat infot saada mitokondriaalse DNA (mtDNA) analüüsidest, sest selle osa pärilikust materjalist pärib järglane ainult emalt. Uuringu põhjal (Sild et al., 2019b) on tori hobuste mitokondriaalne varieeruvus suurem kui teistel Eestis aretatud tõugudel. Joonisel 1 on venni diagrammil esitatud eesti, eesti raskeveo hobuse ja tori hobusetõugude võrdlus ja tõuge eristavate emaliinide osa. Uuritud 110 hobusel tuvastati kokku 38 mtDNA varianti ehk haplotüüpi. Mutatsioonide toimumist arvesse võttes grupeerusid need 38 mtDNA haplotüübid 20 haplogruppi ehk evolutsiooniliselt võiks rääkida 20 kaugemast eellasliinist. Erinevatest emapoolset päritolu markeerivatest haplotüüpidest olid 63% harva esinevad, mida tuvastati vaid ühel või kahel hobusel. Suurim arv harva esinevaid variante leiti tori tõugu hobustel.
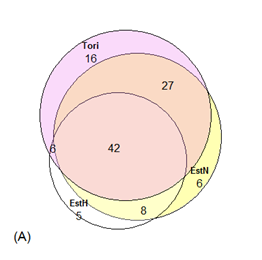
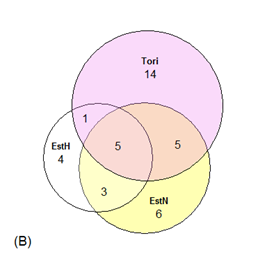
Joonis 1. Mitokondriaalse DNA varieeruvus tori (roosa ring), eesti hobuse (EstN, kollane) ja eesti raskeveohobusel (EstH, valge) tõugude kaupa. Ühiste (ringide kattuvad alad) ja erinevate emaliinidega hobuste (A) ning ühiste ja erinevate haplotüüpide (B) osahulgad. Nii tori kui eesti tõust analüüsiti 40 ning eesti raskeveotõust 30 kolme eellaspõlvkonna alusel omavahel mittesuguluses hobust (Sild et al., 2019b)
Kolmest tõust kokku 42 hobust (38%) kandsid mõnda viiest (13%) tõugude kaupa kattuvast mtDNA haplotüübist. Tori ja eesti tõu erinevus väljendus vastavalt 14 ja 6 erinevas (kolme tõu kontekstis unikaalses) haplotüübis vastavalt 25st ja 19st tuvastatud haplotüübist nendes tõugudes ning ühisosa 10 haplotüübis, mida kandsid kokku 69 (86%) tori ja/või eesti tõugu kuuluvat hobust. Seega on tori ja eesti hobuste ühisosa spetsiifiliselt emapoolset geneetilist tausta hinnates üsna suur. Eesti raskeveohobuste väikesearvulises tõus tuvastati väiksem arv (13) emaliine.
2021. aastal valminud uuringusse olid kaasatud 190 hobuse (eesti, eesti raskeveo, tori (TA, TB, VT), trakeeni, hannoveri ja eesti sporthobuste tõugu) proovid. Genotüpiseerimiseks kasutati hobuste tihedat geenikiipi (Affymetrix Axiom Equine HD Array), kokku 670796 SNPd[1]. Nimetatud tihe geenikiip tagab suurima hobusetõugudel teadaoleva genoomse variatsiooni ja on seetõttu sobivaim limiteeritud arvu proovide juures. Uuringu tellija avalikustas hobuste tõulise kuuluvuse pärast klasteranalüüside läbiviimist. Geneetilistes analüüsides kasutati kokku 222 757 genoomiüleselt määratud SNPd.
Kas eristuvad populatsioonid on tori tõus olemas?
Uuringuküsimusele „Kas eristuvad alampopulatsioonid tori tõus on olemas?“ vastamiseks tegime mitmeid populatsioonigeneetikasse puutuvaid analüüse, et välja selgitada, millise populatsioonisisese struktuuri moodustavad uuringusse kaasatud hobused, st kuidas hobused, hoolimata nende tõuraamatulisest kuuluvusest, tegelikult grupeeruvad ehk mitu geneetilist klastrit moodustub 190 kuude tõugu kuuluva hobuse alusel. Samuti uuriti, kui suur on erinevate tõugude ja tõusiseste gruppide omavaheline distants või vastupidi, kui sarnased nad on. Võtsime arvesse nii tõusisese alampopulatsiooni eristatavust (diferentseerumist geneetiliste markerite alusel) kui eristatavat (alam)populatsiooni puudutavat aretusmeetodit – kooskõlas seadustega tuleks erineva nimega tähistada alampopulatsiooni, mis on puhasaretuse (sisearetuse) teel loodud aretusliin.
Genoomiandmete põhjal jaotusid anonüümsena analüüsitud 190 hobust vastavalt oma geneetilisele profiilile nelja erineva suurusega geneetilisse klastrisse. Vastavalt klaster- ja Bayesi statistilisele analüüsile (etteantud mudeli järgi) moodustasid indiviidid erineva suurusega populatsioonid: 26, 26, 43 ja 95 hobust. Varieeruv oli ka statistiline tõepära (38-99%), millega indiviidid teatud populatsiooni juurde klasterdusid (klastrikuuluvuse tõenäosusega). Suur hulk hobuseid näitas ka suhteliselt ühesuurust erinevaisse populatsioonidesse kuulumist, seega kirjut ehk paljukomponentset eellaspäritolu.
Klasteranalüüsi tulemuste võrdlemisel indiviidide tõukuuluvuse andmetega selgus, et väiksemad ja homogeensema genofondiga kaks klastrit moodustusid 100% üks eesti ja teine eesti raskeveo hobuste alusel. Suuruselt järgmine, 43 indiviidiga klaster moodustus 41 tori (kokku oli analüüsis 68 tori hobust) ja 2 eesti sporthobuse alusel. Neljandas klastris olid trakeeni, hannoveri, tori ja eesti sporthobused. Tori hobustest klasterdusid sellesse mitmest tõust koosnevasse populatsiooni peamiselt tori TB hobused, kuid nimetatud TB-osa hobused olid geneetiliselt heterogeensed, sh geenikomponentide erineva osakaaluga.
SNPde alusel ilmnenud geneetilist struktuuri aitavad näitlikustada peakomponentanalüüsi (PCA) joonised (joonis 2). PCA analüüsis teisendati hobuste genoomiinfo erinevused ja sarnasused mitmemõõtmelise ruumi distantsideks, mis võimaldab indiviidide esitamist mitmel 2D graafikul, nii et indiviidide omavaheline paiknemine väljendab geneetiliste erinevuste suurust. Samasse tõugu kuuluvate hobuste keskmine sugulus on suurem kui erinevatesse tõugudesse kuuluvate hobuste vahel ning seetõttu on sama tõu hobused oodatavalt üksteisega geneetiliselt sarnasemad, moodustades genotüüblise läheduse tõttu teistest tõugudest pärit hobustest eristuvaid klastreid.
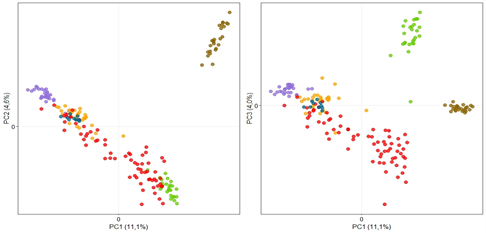
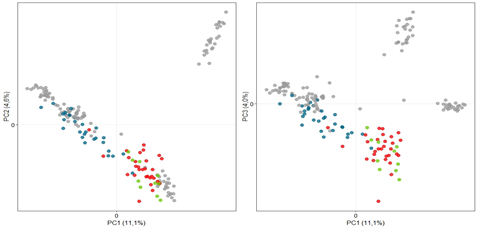
Joonis 2. Uuringusse kaasatud 190 hobuse peakomponentanalüüsi tulemused 222757 SNP alusel.
Esitatud joonised näitavad varieeruvust peakomponentanalüüsi kolme esimese komponendi alusel: x-teljel on peakomponent (PC) 1 ja y-teljel PC 2 (vasakpoolne joonis) või PC 3 (parempoolne joonis). Igale täpile vastab üks indiviid. Täpikese värvus näitab tõu ja tõuraamatuosa kuuluvust.
Ülemisel joonisel (A ja B): tori hobune – punane; eesti hobune – roheline; eesti raskeveohobune – pruunikas; hannoveri hobune – sinine; eesti sporthobune – kollane; trakeeni hobune – lilla.
Alumisel joonisel (C ja D) on esile toodud tori tõuraamatu erinevatesse osadesse kuuluvad hobused: tori TA – punane; tori TB – sinine; tori VT – roheline.
Joonistel on näha, et tori hobused paiknevad võrreldes eesti ja eesti raskeveohobustega hajusamalt – tori hobused on tõusiseselt mitmekesisemad, hõivates ala eesti hobustest kuni trakeenini (homogeenseim tõug). Tori TA ja VT hobused on kattuval alal – sellist paigutust toetas diferentseerumisindeks (FST=0) – TA ja VT alampopulatsioonid geneetiliselt ei eristunud.
Tori aretussuuna TB hobused paiknevad joonisel nii, et katavad TA ja VT hobuste ning hannoveridega segunenud sporthobuste klastri vahelise ala. Väiksem osa analüüsitud TB hobustest on segunenud TA-VT ja suurem osa teise klastriga. Selline TB hobuste paigutus oli kooskõlas teiste populatsioonidevahelisi erinevusi mõõtvate näitajatega. TB hobused olid suhteliselt madala difereerumisindeksiga TA ja VT alampopulatsioonidest, kuid veelgi enam eesti sporthobuse ja hannoveri tõust. Tori aretussuuna hobuste puhul on oluline asjaolu, et TB hobused on üksteisest erinevamad kui mistahes teise analüüsitud tõu hobused üksteisest. Keskmine TB indiviidide paariviisiline sarnasuskoefitsient oli võrreldav vaid sporthobuste sama näitajaga, kus välisaretus on tavapärane. Genoomiandmete analüüsi tulemused näitavad, et tori TB hobused tori tõu sisese grupina ei moodusta geneetiliselt ühtlikku alampopulatsiooni.
Genoomiandmetele tuginedes saab teha järgmise kokkuvõtte:
- tori tõus tõusiseseid alampopulatsioone eristuvate, puhtatõuliste aretusliinide mõistes ei tuvastatud;
- tori tõusiseste erinevate aretussuundade mõttes analüüsitud alampopulatsioonid (valimid TA, TB ja VT hobustest) on geneetiliselt sarnased – nende omavaheline eristumine on madal;
- TB hobuste puhul on eristumine veelgi madalam eesti sporthobustest ning analüüsitud komponenttõugudest (trakeen, hannover) võrreldes TA ja VT hobustega;
- TB hobused varieerusid tori ja soojavereliste geenikomponentide proportsioonierinevuste poolest suures ulatuses;
- tori TA, TB ja VT hobused jaotusid mitte rohkem kui kahte erinevasse geneetilisse populatsiooni, üldistavalt klasterdusid enamik TA ja VT ning vähemus TB hobuseid tori populatsiooni ja enamik TB hobuseid soojavereliste hobuste populatsiooni;
- tori TA hobuste ja VT hobuste vahel geneetiline eristumine puudus – VT tõuraamatu hobused tõusisest alampopulatsiooni geeniandmete alusel ei moodustanud.
Genoomiandmete kasutamine tõu säilitamise meetmete väljatöötamiseks on üks komponentidest, millele tugineda. Tõu säilimiseks on vaja, et geneetiline materjal oleks mitmekesine ehk tõus olev geneetiline variatsioon ei väheneks.
[1]SNP – üksiknukleotiidne polümorfism genoomis (ingl. Single Nucleotide Polymorphism). Kindlas DNA-punktis oleva üksiku aluspaari vahetusvarieeruvus populatsioonis
Viidatud allikad
Sild, E.; Rooni, K.; Värv, S.; Røed, K.; Popov, R.; Kantanen, J.; Viinalass, H. (2019a). Genetic diversity of Estonian horse breeds and their genetic affinity to northern European and some Asian breeds. Livestock Science, 220, 57−66. DOI: 10.1016/j.livsci.2018.12.006.
Sild, E.; Värv, S.; Kaart, T.; Kantanen, J.; Popov, R.; Viinalass, H. (2019b). Maternal and paternal genetic variation in Estonian local horse breeds in the context of geographically adjacent and distant Eurasian breeds. Animal Genetics, 50 (6), 757−760. DOI: 10.1111/age.12835.
Uuringu „Tori hobusetõu ja selle alampopulatsioonide ning teiste kohalike Eesti hobusetõugude geneetiline analüüs ning nende sarnasuse või eristatuse võrdlus“ lõpparuanne. 2021. Autorid: S. Värv, E. Sild, T. Kaart, K. Jõgi, M. Vallas, H. Viinalass.
Uuringu „Tori hobusetõu analüüs põlvnemisandmete ja DNA markerite põhjal” lõpparuanne. 2013. Autorid: K. Rooni, E. Sild, S. Värv, H. Viinalass.




